Die blutbasierte Analyse von zirkulierenden Tumorzellen oder von Nukleinsäuren, die vom Tumor stammen, wird als Liquid Biopsy (Flüssigbiopsie) bezeichnet. Das Hauptziel dieser Methode ist die Analyse zellfreier DNA (“cell-free DNA”, cfDNA), die von nekrotischen und apoptotischen Zellen in den Blutkreislauf abgegeben wird. Erhöhte Werte von cfDNA können bei Patientinnen und Patienten mit Krebs und anderen Erkrankungen festgestellt werden. Da nur ein kleiner Teil der zirkulierenden DNA aus dem Tumor stammt (“circulating tumor DNA”, ctDNA), sind hochsensible Nachweisverfahren erforderlich.
Die Anwendungsmöglichkeiten von Liquid Biopsys sind vielfältig und weisen gegenüber der konventionellen Gewebeanalyse deutliche Vorteile auf: Unter anderem basiert die Analyse von Liquid Biopsys auf einer einfachen Blutentnahme und kann daher leicht wiederholt werden.
Anwendungsbereiche von Liquid Biopsys sind:
- Überwachung von Tumorerkrankungen
- Überwachung des Therapieansprechens
- Stratifizierung von Patientinnen und Patienten sowie deren Behandlungsauswahl
- Nachweis einer minimalen Resterkrankung
- frühzeitige Erkennung und Profiling einer Therapieresistenz
Wir bieten verschiedene Liquid-Biopsy-Produkte an, um zuverlässige und genaue Einblicke in die zellfreie DNA zu erhalten.
CeGaT ist der beste Partner für Ihr Sequenzierprojekt
Unser Engagement für Sie
Schnelle Bearbeitung
Bearbeitungszeit
≤ 15 Werktage
Hohe Qualität
Höchste Genauigkeit bei allen Prozessen
Sichere Datenlieferung
Sichere Bereitstellung der sequenzierten Daten über hausinterne Server
Sichere Aufbewahrung
Sichere Proben- und Datenaufbewahrung nach Projektabschluss
Unser Service
Uns ist eine umfangreiche und erstklassige Projektbegleitung wichtig − von der Auswahl des passenden Produktes bis zur Auswertung der Daten. Jedes Projekt wird von einer engagierten Wissenschaftlerin oder einem engagierten Wissenschaftler begleitet, so dass Ihnen während des gesamten Projektverlaufs eine Ansprechpartnerin oder ein Ansprechpartner zur Seite steht.
Unser Service umfasst:
- Ausführliche Projektberatung
- Produktauswahl abgestimmt auf Ihr Projekt
- Umfangreiche bioinformatische Auswertung Ihrer Daten
- Abschließender und detaillierter Projektbericht mit Informationen zur Probenqualität, Sequenzierparametern, bioinformatischen Analysen und Ergebnissen
Profitieren Sie von unserem hervorragenden Support und unseren akkreditierten Arbeitsabläufen.
Unser Produktportfolio für Liquid Biopsy
Wir bieten verschiedene Liquid-Biopsy (LB)-Produkte für eine Vielzahl von Forschungsfragen an. Wenn Sie LB Target wählen, entscheiden Sie sich für einen ddPCR-Ansatz (“droplet digital PCR”, ddPCR). Dieser Ansatz eignet sich besonders für die Überwachung einzelner Genvarianten. LB Focus zielt auf ausgewählte krebsassoziierte Gene ab. Bei diesem Next-Generation-Sequencing-basierten Ansatz wird ein von CeGaT entwickeltes Gen-Panel verwendet. Wenn Sie sich für LB Exploratory entscheiden, erhalten Sie ein umfassendes genomisches Profiling von cfDNA, unter Verwendung des TSO500 ctDNA Assays von Illumina. Für LB Flex kann entweder das gesamte Exom oder ein vordefiniertes Set an Genen analysiert werden.
Wünschen Sie zusätzlich zu den beinhalteten Leistungen bioinformatische Analysen Ihrer Daten? Jedes unserer Produkte kann durch weitere Dienstleistungen ergänzt werden. Wir beraten Sie gerne.
LB Target | LB Focus | LB Exploratory | LB Flex |
Spezies | Spezies | Spezies | Spezies |
Ausgangsmaterial | Ausgangsmaterial | Ausgangsmaterial | Ausgangsmaterial |
Ziel | Ziel | Ziel | Ziel |
Technologie | Technologie | Technologie | Technologie |
Bioinformatik-Pipeline | Bioinformatik-Pipeline | Bioinformatik-Pipeline | Bioinformatik-Pipeline |
Duplex-UMIs | Duplex-UMIs | Duplex-UMIs | Duplex-UMIs |
Variantentyp | Variantentyp | Variantentyp | Variantentyp |
Beinhaltete Leistungen | Beinhaltete Leistungen | Beinhaltete Leistungen | Beinhaltete Leistungen |
Bioinformatik
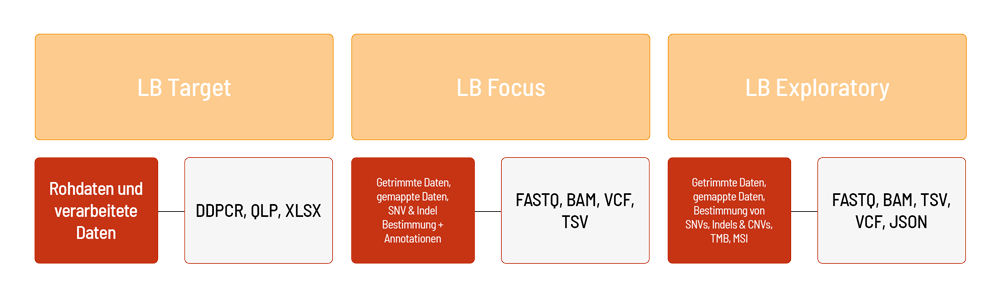
Für LB Target sind die Ergebnisse der ddPCR als Rohdaten (DDPCR, QLP-Dateien) und verarbeitete XLSX-Dateien (Kopien/µl, Tröpfchenanzahl usw.) verfügbar. Zusätzlich zu den Daten wird ein Projektbericht erstellt (PDF-Datei).
Für LB Focus werden die sequenzierten Rohdaten (FASTQ-Datei) automatisch auf Basis unserer eigenen Bioinformatik-Pipeline verarbeitet. Die Analyse von LB Focus beinhaltet das Alignment der getrimmten Daten (BAM-Datei), sowie die Bestimmung und Annotation von Einzelnukleotid-Varianten (Single nucleotide variants, kurz SNVs) und kleine Insertionen und Deletionen (kurz Indels) (VCF- und TSV-Dateien). Zusätzlich zu den Daten wird ein Projektbericht erstellt (PDF-Datei).
Für LB Exploratory werden die sequenzierten Rohdaten (FASTQ-Datei) automatisch auf Basis der TSO500-Pipeline (Illumina) verarbeitet. Die Analyse für LB Exploratory umfasst das Alignment der getrimmten Sequenzierdaten (BAM-Datei), die Bestimmung und Annotation von Einzelnukleotid-Varianten (Single nucleotide variants, SNVs) und kleinen Insertionen und Deletionen (Indels) sowie von Fusionen (VCF-, TSV- und JSON-Dateien). Darüber hinaus werden Kopienzahlveränderungen (Copy number variations, CNVs) (VCF-Datei) und der MSI/TMB-Status ausgewertet (TSV-Datei). Zusätzlich zu den Daten wird ein Projektbericht erstellt (PDF-Datei).
Für LB Flex werden die Rohdaten automatisch mit der DRAGEN Bio-IT Plattform verarbeitet. Wir bieten verschiedene Level bioinformatischer Analysen für LB Flex an. Das Standardlevel ist Level 1. Mit steigendem Bioinformatiklevel werden mehr Daten geliefert. Alle höheren Level beinhalten dabei die Daten der vorherigen Level. Zusätzlich zu den Daten und unabhängig vom Analyselevel wird ein Projektbericht verfasst.
Level 1:
- Demultiplexing und Adapter-Trimming der Sequenzierdaten (FASTQ-Datei)
- Metriken (CSV-Datei)
- MultiQC-Bericht (HTML-Datei)
Level 2:
- Mapping der Sequenzierdaten (BAM-Datei)
- Metriken (CSV-Datei)
Level 3:
- Bestimmung von Einzelnukleotid-Varianten (Single nucleotide variants, kurz SNVs) und kleinen Insertionen und Deletionen (kurz Indels) (VCF-Datei)
- Metriken (CSV-Datei)
Level 4:
- Annotation von SNVs und Indels (JSON- und TSV-Datei)
Technische Information
Bei CeGaT wird die ddPCR mit dem QX200 Droplet Digital PCR System (Bio-Rad) durchgeführt. Die Paired-End-Sequenzierung (2 x 100 bp) wird mit den Sequenzierplattformen von Illumina durchgeführt. Wenn Sie andere Sequenzierparameter benötigen, lassen Sie es uns gerne wissen! Wir können Ihnen weitere Lösungen anbieten.
Genverzeichnis für LB Focus, LB Exploratory und LB Flex (Panel)
Weitere Informationen zu Liquid Biopsy
Die Analyse der zellfreien Tumor-DNA hat gegenüber der herkömmlichen Gewebeanalyse mehrere entscheidende Vorteile:
- Sie stellt immer ein aktuelles Bild der Erkrankung dar.
- Sie gibt Aufschluss über die Heterogenität des gesamten Tumors und nicht nur eines kleinen Teils davon.
- Sie basiert auf einer einfachen Blutentnahme. Das bedeutet, dass die Untersuchung patientenfreundlich, nicht-invasiv und leicht zu wiederholen ist.
Diese Vorteile eröffnen ein weites Einsatzspektrum. Im Folgenden werden wir Ihnen zwei mögliche Anwendungsbeispiele geben.
Für die Durchführung einer klinischen Studie, z. B. für ein neues Medikament, möchte man
- die möglichen Wirkungen ermitteln,
- Patientinnen und Patienten, die auf das Medikament ansprechen, identifizieren,
- den dahinterstehenden Reaktionsmechanismus verstehen,
- mögliche Biomarker identifizieren,
- die Entwicklungsdynamik des Tumors verfolgen
- und Resistenzmechanismen aufdecken.
Für diese Zwecke haben wir unser Liquid-Biopsy-Exploratory-Produkt entwickelt. Dieses Produkt umfasst die Analyse von mehr als 520 krebsassoziierten Genen sowie immunonkologischen Biomarkern. Es werden so genannte Unique Molecular Identifiers, kurz UMIs, verwendet.
Zusätzlich sequenzieren wir mit einer sehr hohen Sequenziertiefe. Somit kombiniert Liquid Biopsy Exploratory eine umfassende genomische Profilerstellung aus dem Plasma mit einer hohen Sensitivität.
Ein weiteres Anwendungsgebiet von Liquid Biopsy ist das Monitoring einer Tumorerkrankung und deren Therapieansprechen. Liquid Biopsys können eingesetzt werden, wenn der Tumor der erkrankten Person und die Treibermutationen bereits analysiert und bekannt sind und die Person eine stabile Erkrankung oder sogar ein vollständiges Ansprechen auf die Therapie zeigt, aber das Risiko eines Rückfalls besteht. Mit Liquid Biopsys kann das mögliche Auftreten einer Resterkrankung oder von Rezidiven so früh wie möglich erkannt werden. Zu diesem Zweck ist unser Produkt Liquid Biopsy Focus gut geeignet. Die Analyse konzentriert sich auf eine relativ kleine Anzahl ausgewählter Gene. Nichtsdestotrotz ist eine hohe Sensitivität erforderlich. Daher verwenden wir Duplex-UMIs in Kombination mit einer hohen Sequenziertiefe. Dadurch können wir Varianten mit einer Allelfrequenz von bis zu 0,25 % nachweisen.
Dies waren nur zwei mögliche Anwendungsbeispiele. Wir wissen jedoch, dass jedes Projekt und seine Anforderungen unterschiedlich sind. Deshalb bieten wir verschiedene Dienstleistungen für die Analyse von Liquid Biopsys an und beraten Sie gerne bei der Suche nach der optimalen Strategie für Ihr Projekt. Wir sind Ihr kompetenter Partner bei der Überführung von Liquid-Biopsy-Assays in die klinische Praxis.
Video: Nutzen Sie die vielfältigen Möglichkeiten von Liquid Biopsy (EN)
Downloads
Kontaktieren Sie uns
Sie haben noch Fragen oder Interesse an unserem Service? Treten Sie gern mit uns in Kontakt. Wir werden uns schnellstmöglich um Ihr Anliegen kümmern.
Starten Sie Ihr Projekt mit uns
Gerne beraten wir Sie zu unseren Sequenzierdienstleitungen und erarbeiten mit Ihnen gemeinsam die beste Lösung, die auf Ihre klinische Studie oder Forschungsprojekt abgestimmt ist.
Bitte geben Sie, falls möglich, folgende Probeninformationen an: Ausgangsmaterial, Anzahl der Proben, bevorzugte Option für die Vorbereitung der Library, bevorzugte Sequenziertiefe und gewünschte bioinformatische Analysestufe.