Die personalisierte Krebsbehandlung gewinnt immer mehr an Bedeutung. Die Entwicklung eines personalisierten Krebsimpfstoffs ist eine vielversprechende Strategie zur Behandlung von Krebs.
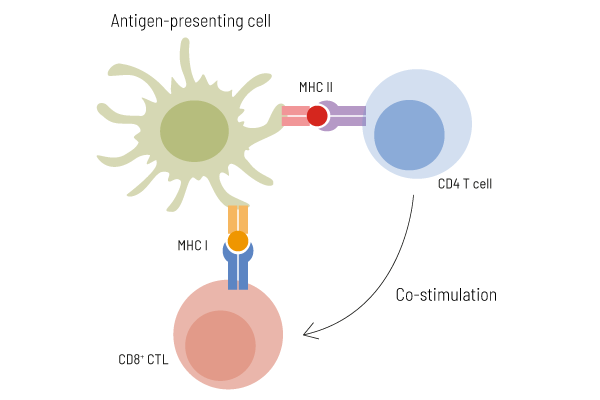
Körperzellen präsentieren auf ihrer Oberfläche permanent sogenannte Peptide, die den aktuellen Zustand der Zelle anzeigen. Immunzellen können erkennen, ob eine Zelle über normale oder abnormale Peptide verfügt und nutzen diese Informationen, um defekte Zellen zu erkennen und zu eliminieren. Abnormale Peptide, sogenannte Neoantigene, entstehen durch Mutationen der DNA in den Tumorzellen und sind Zielobjekte für die Therapie mittels personalisierter Vakzine.
CeGaTs CancerNeo® analysiert das Tumorexom einer Patientin oder eines Patienten, um tumorspezifische (somatische) Mutationen zu erkennen, die HLA-Typen zu identifizieren und Neoantigene vorherzusagen. Die Expression dieser Neoantigene wird durch die Sequenzierung der gesamten RNA (Transkriptom) aus derselben Tumorprobe bestätigt. Damit liefert CancerNeo® die notwendigen Erkenntnisse für die Entwicklung personalisierter Krebsimpfstoffe und ist ein leistungsfähiges Instrument, um die Reaktion des Immunsystems auf Krebszellen zu verstärken.
Sie sind in Deutschland versichert? Unsere Kolleginnen und Kollegen vom Zentrum für Humangenetik Tübingen beraten Sie gerne!
Was wir Ihnen mit diesem Service bieten
Unser Versprechen an Sie
Service Details
- Exom-Sequenzierung von Tumor- und Normalgewebe mit CeGaTs ExomeXtra®
- Detaillierte Bewertung der somatischen Varianten, die in mehr als 700 tumorrelevanten Genen und Fusionen in mehr als 30 Genen entdeckt wurden
- Medizinischer Bericht mit:
-
- Varianten mit therapeutischer Relevanz
- Behandlungsmöglichkeiten basierend auf somatischen Varianten
- Analyse der Tumormutationslast (TMB), der Mikrosatelliteninstabilität (MSI) und Berechnung des HRD-Scores
- Erkennung von therapierelevanten Kopienzahlveränderungen (copy number variants, CNV)
- Auflistung aller in Frage kommenden Medikamente mit EMA und/oder FDA-Zulassung für deren Anwendungsoption entsprechende Biomarker im Tumor nachgewiesen werden konnten
- Bestimmung von pharmakogenetisch relevanten Keimbahnvarianten, die die Verstoffwechselung bestimmter Tumormedikamente oder Narkosemittel beeinflussen
- Erfassung von Hinweisen auf CHIP (Klonale Hämatopoese von unbestimmtem Potential)
-
- Sequenzierung des Tumor-Transkriptoms
- HLA-Typisierung
- Vorhersage von Peptidepitopen (Neoepitope) der HLA-Klasse I, die tumorspezifische Varianten überspannen
- Auswahl der relevantesten Neoepitope, die an HLA-Klasse I und HLA-Klasse II binden
- Zweiter medizinischer Bericht mit ausgewählten Peptiden für die Impfstoffentwicklung
Optional:
- RNA-basierte Fusions-Transkript-Analyse (CancerFusionRx®) mit über
150 Genen für den Nachweis von Fusionen und über 120 Exon-Exon spezifische Anreicherungen mit bekannten Bruchpunkten — mehr erfahren - Immunhistochemische (IHC)-Analysen: PD-L1, CAR-T-Zellen-Panel, HLA-Klasse I und HLA-Klasse II-Färbung (extern) — mehr erfahren
- MGMT-Promoter-Methylierungsanalyse
Beispielbefund
Unsere Standaranforderungen für Proben
Normales Gewebe
- 1–2 ml EDTA-Blut (empfohlene Probenart), oder
- Genomische DNA (1–2 µg)
Tumorgewebe
Tumorgehalt mindestens 20 %
- FFPE-Tumorblock (min. Gewebegröße 5x5x5 mm) (empfohlene Probenart)
- FFPE-Tumorgewebe-Objektträger (min. 10 Schnitte 4-10 µm, Gewebegröße 5×5 mm)
- Genomische DNA (> 200 ng)
- frisch gefrorenes Tumorgewebe
- 3x 10 ml cfDNA-Röhrchen für Flüssigbiopsie
Hier finden Sie weitere Informationen zum sicheren Versand Ihrer Probe.
Weitere Probenmaterialien
Andere Probenmaterialien sind auf Anfrage möglich. Bitte beachten Sie: Bei unzureichender Probenqualität oder Tumorgehalt kann die Analyse fehlschlagen.
Wenn Sie mehr als eine Option für Tumorproben haben, wenden Sie sich bitte an unseren Diagnostik Support.
Wir unterstützen Sie gerne bei der Auswahl der optimalen Probe für Ihre Patientinnen und Patienten. Für höchste Genauigkeit benötigen wir Tumor- und Normalgewebe für unser somatisches Tumordiagnostik-Panel.
Das macht unseren CancerNeo® Service besonders
Die Entwicklung eines personalisierten Krebsimpfstoffs erfordert höchste Präzision bei der Identifizierung der somatischen Varianten sowie bei der anschließenden Vorhersage und Auswahl von Neoantigenen, die im Impfstoffdesign verwendet werden sollen. CeGaTs CancerNeo® identifiziert somatische Veränderungen durch eine vergleichende Analyse von Tumor- und Normalgewebe. Die Analyse basiert auf CeGaTs ExomeXtra®, der bestmöglichen Exom-Anreicherung, und ist die präziseste Methode zur Identifikation somatischer Varianten. Die Transkriptom-Sequenzierung identifiziert hoch exprimierte potenzielle Zielmoleküle für das Impfstoffdesign und ist Teil unseres Selektionsprozesses, der auf unserer langjährigen Erfahrung beruht. Damit ermittelt CancerNeo® verlässlich die vielversprechendsten Neoantigene.
Option auf zusätzliche Panel-Sequenzierung
Die im Rahmen von CancerNeo® durchgeführte Analysie basiert auf einer Exom-Sequenzierung, da für die Vorhersage von Neoantigenen ein Verständnis aller somatischen Mutationen notwendig ist. Unsere spezialisierte Anreicherung des somatischen Tumorpanels, auf welchem unser diagnostischer Service CancerPrecision® basiert, ermöglicht die fokussierte Analyse tumorassoziierter Gene und ausgewählter Translokationen. Diese werden daher in viel höherer Auflösung sequenziert. Die zusätzliche Panel-Sequenzierungs-Option stützt den medizinischen Bericht zur Unterstützung der Behandlungsentscheidung (CancerPrecision®) auf die Sequenzierungsdaten aus der spezialisierten Panel-Anreicherung. Auf der anderen Seite basiert der medizinische Bericht für vorhergesagte Neoantigene zur Entwicklung personalisierter Impfstoffe auf den gesamten Exom-Sequenzierdaten. Der CancerPrecision®-Bericht profitiert von der höheren Sequenzierungsauflösung und der Möglichkeit, die ausgewählten, durch die Panel-Sequenzierung angereicherten Translokationen nachzuweisen.
Was ist eine Neoantigen-Vorhersage?
Neoepitope können von CD8+ zytotoxischen T-Zellen (CTL) und von CD4+ T-Helferzellen erkannt werden. Neoepitope, die von CD8+ T-Zellen erkannt werden, sind typischerweise 8-12 Aminosäuren lang und werden von HLA-Klasse-I-Molekülen präsentiert. Neoepitope, die von CD4+ T-Helferzellen erkannt werden, sind dagegen üblicherweise länger (13-18 Aminosäuren) und werden von HLA-Klasse-II-Molekülen präsentiert. Für eine erfolgreiche Immuntherapie sollte die Auswahl der Neoantigene kurze und lange Peptide umfassen, um sowohl CD4+- als auch CD8+-T-Zellen zu aktivieren. Die Immunreaktionen der Krebserkrankten können während der Krebsimpfung durch eine auf Durchflusszytometrie basierende Analyse der Neoepitop-spezifischen T-Zell-Aktivierung überwacht werden.
Unsere Krebsimmunologie-Expertinnen und -Experten verwenden in-silico Algorithmen, die auf Exom-Sequenzierung und HLA-Typisierung basieren, um bis zu 12 geeignete Neoantigen-Epitope vorherzusagen und auszuwählen, die für Patientinnen und Patienten individualisiert sind. Die ausgewählten Peptide werden nicht nur zur Aktivierung zytotoxischer T-Zellen, sondern auch von T-Helferzellen vorhergesagt. Daher sind neben kurzen Peptiden (8-12 Aminosäuren), die potenziell an HLA-Klasse-I-Moleküle binden können, auch lange Peptide (13-18 Aminosäuren) enthalten, die potenziell an HLA-Klasse-II-Moleküle binden können. Um die Expression der ausgewählten Neoepitope zu bestätigen, werden parallel dazu Transkriptomdaten durch RNA-Sequenzierung der Tumorprobe erstellt. In Fällen, in denen keine RNA verfügbar ist, werden Informationen über die Expression der Neoepitope aus Proteinexpressionsdatenbanken verwendet.
Immunhistochemie-Analyse (IHC)
Zur Vervollständigung unserer genetischen Diagnostik bieten wir an, in Zusammenarbeit mit Partnerlaboren immunhistochemische Analysen der Tumorprobe zu organisieren. Die pathologischen Untersuchungsberichte leiten wir nach Fertigstellung weiter.
PD-L1
Der Nachweis der Expression des Programmed death-ligand 1 (PD-L1) im Tumorgewebe ist wichtig für die Auswahl von Patientinnen und Patienten, die (am meisten) von einer Immuntherapie wie Pembrolizumab profitieren können.
Analyse der Methylierung des MGMT-Promotors
Der Nachweis der Methylierung des MGMT-Promotors im Tumorgewebe ist für Gliom-Patientinnen und -Patienten wichtig, da er ein potenzieller Biomarker für die Empfindlichkeit gegenüber alkylierender Chemotherapie, einschließlich Temozolomid (TMZ), ist.
HLA Klasse I und HLA Klasse II
HLA-Moleküle präsentieren den T-Zellen Tumorantigene. Die Expressionsanalyse von HLA-Molekülen ist ein hilfreiches Werkzeug, um Ihre immuntherapeutische Strategie maßzuschneidern.
CAR T-Zell-Panel (GD2, EGFR, IL13Ralpha, CD276, HER2, PSMA, ROR1, CD47)
Das CAR-T-Zell-Panel detektiert acht verschiedene Zielantigene von CAR-T-Zell-Therapien in der klinischen oder präklinischen Entwicklung.
CancerFusionRx®
RNA-basierte Identifizierung von Fusionstranskripten
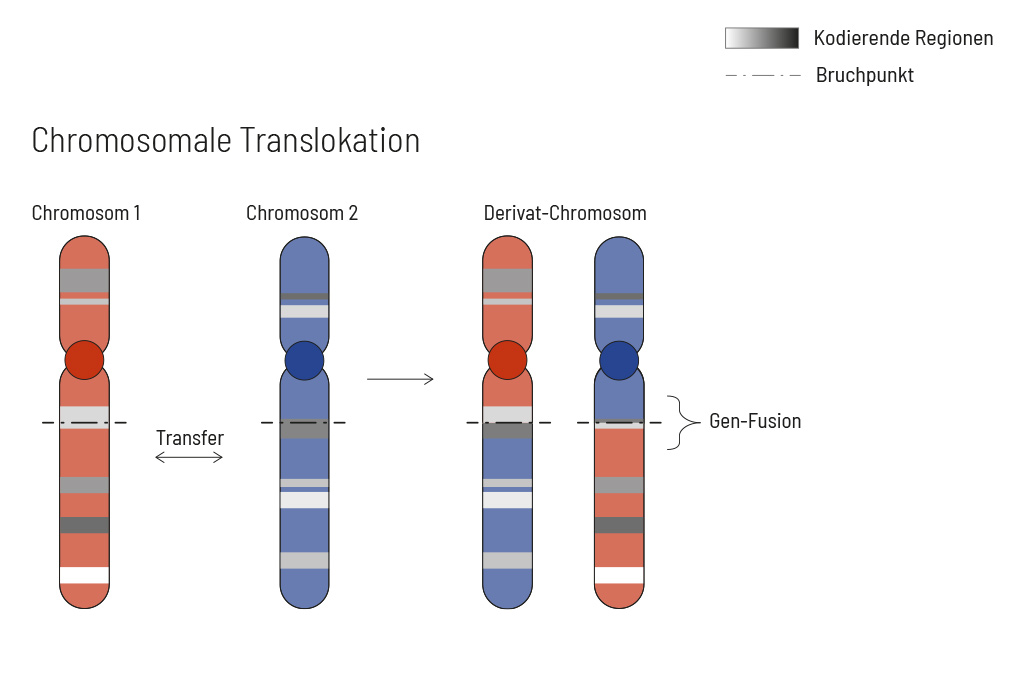
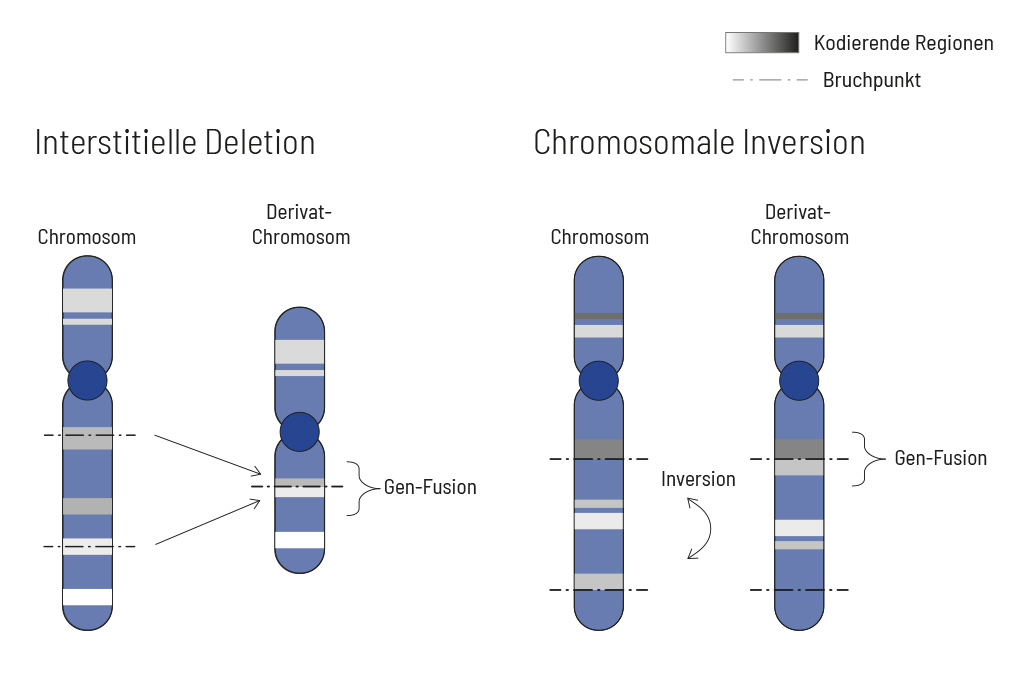
Chromosomale Veränderungen treten häufig und unabhängig von der Tumorentität auf. Infolgedessen kann es zu Genfusionen im Tumorgenom kommen. Fusionen sind oftmals starke Treiber der Tumorerkrankung und daher für Behandlungsentscheidungen sehr wichtig. Herkömmliche, auf PCR-Technologie basierende Methoden, können eine Fusion nicht nachweisen, wenn der Fusionspartner nicht bekannt ist (dies ist bei NTRK-Fusionen häufig der Fall). Selbst Analysen des gesamten Transkriptoms sind nicht sensitiv genug, insbesondere wenn der Tumorgehalt niedrig ist. Um alle bekannten und zuvor beschriebenen, sowie neuartigen Genfusionen mit einer therapeutischen Option nachzuweisen, haben wir eine gezielte Anreicherung auf RNA-Basis entwickelt.
Das Design umfasst aktuell mehr als 150 Gene für den Nachweis von Fusionen und beinhaltet über 120 Exon-Exon spezifische Anreicherungen mit bekannten Bruchpunkten. Diese Methode ist den DNA-basierten Methoden und auch Ansätzen auf Transkriptom-Basis überlegen. Wir empfehlen dringend, die genetische Tumordiagnostik durch RNA-Anreicherung für Fusionen zu ergänzen, um ein möglichst vollständiges Verständnis der Tumorbiologie zu erhalten.
Downloads
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.