Jeder Tumor ist einzigartig. CancerPrecision® hilft, die klinisch relevanten Veränderungen in Tumoren zu identifizieren und liefert wertvolle Informationen für die Wahl der individuell effektivsten Behandlung für jede Patientin und jeden Patienten.
CancerPrecision® ermöglicht ein optimales molekulargenetisches Tumor-Profiling durch NGS und bildet die Grundlage für eine personalisierte, biomarkerbasierte Krebstherapie.
Unser Ziel ist es, den bestmöglichen therapeutischen Ansatz für jede Patientin und jeden Patienten zu ermitteln. Basierend auf unserer langjährigen Erfahrung in der genetischen Diagnostik haben wir die Tumordiagnostik optimiert. Wir identifizieren somatische Veränderungen, die das Tumorwachstum fördern, für Arzneimittelresistenzen verantwortlich sind und potenzielle therapeutische Ziele darstellen. Mit Hilfe von NGS analysieren wir ein Panel mit mehr als 700 tumorassoziierten Genen und ausgewählten therapierelevanten Fusionen in mehr als 30 Genen. Die gezielte RNA-basierte Fusionsanalyse als zusätzliche Option ermöglicht den Nachweis von Fusionstranskripten mit de-novo– und bekannten Fusionspartnern in mehr als 150 Genen. Varianten in diesen Genen haben einen bedeutenden Einfluss auf die Tumorpathogenese und -progression, sowie auf die Bildung von Metastasen. Im Hinblick auf Immuntherapien bestimmen wir die Tumormutationslast (TMB), die Mikrosatelliteninstabilität (MSI) und das Vorliegen einer Virusinfektion (HPV, EBV). Darüber hinaus bestimmen wir den Status der homologen Rekombinationsdefizienz (HRD), der wichtige Informationen für die Vorhersage des Ansprechens auf PARP-Inhibitoren und eine Platinbasierte Chemotherapie mittels synthetischer Letalität liefert. Innerhalb von 2–3 Wochen werden die Daten in einem umfassenden Befund zusammengefasst, der die behandelnden Ärztinnen und Ärzte dabei unterstützt, eine effiziente Behandlung zu finden.
Sie sind in Deutschland versichert? Unsere Kolleginnen und Kollegen vom Zentrum für Humangenetik Tübingen beraten Sie gerne!
CancerPrecision® ist die erste Wahl zur Charakterisierung des Tumors
1 Basierend auf einer qualitativ hochwertigen Probe mit 20 % Tumorgehalt zum Nachweis einer somatischen heterozygoten Variante.
Unser Versprechen an Sie
Service Details
- Vollständige Sequenzierung und Analyse von mehr als 700 Genen und Fusionen in über 30 Genen — mehr erfahren
- Hohe Sequenzier-Coverage zum Nachweis therapierelevanter subklonaler Varianten: 500-1.000x
- Sensitivität: > 96 %*; Spezifität: > 99,9 %
- Analyse der Tumormutationslast (TMB), der Mikrosatelliteninstabilität (MSI) und Virusinfektionen (HPV, EBV) — wichtige Biomarker für Immuntherapien — mehr erfahren
- Berechnung des Scores der homologen Rekombinationsdefizienz (HRD) — ein wichtiger Biomarker für PARP-Inhibition — mehr erfahren
- Erkennung von Einzelnukleotidvarianten (SNVs), Insertionen und Deletionen (Indels), Translokationen und Kopienzahlveränderungen (copy number variants, CNVs) — mehr erfahren
- Neben therapierelevanten somatischen (tumorspezifischen) Mutationen werden auch krankheitsverursachende und therapierelevante Keimbahnvarianten berichtet
- Bestimmung von ausgewählten pharmakogenetisch relevanten Keimbahnvarianten, die die Verstoffwechselung bestimmter Krebsmedikamente beeinflussen
- Auflistung aller in Frage kommenden Medikamente mit EMA und/oder FDA-Zulassung, für deren Anwendungsoption entsprechende Biomarker im Tumor nachgewiesen werden konnten — mehr erfahren
- Erfassung von Hinweisen auf CHIP (Klonale Hämatopoese mit unbestimmtem Potenzial)
Optional:
- RNA-basierte Fusionstranskript-Analyse von Tumor-RNA zur Analyse von > 150 Genen (CancerFusionRx®) — mehr erfahren
* Basierend auf einer qualitativ hochwertigen Probe mit 20 % Tumorgehalt zum Nachweis einer somatischen heterozygoten Variante.
Beispielbefund
Unsere Standaranforderungen für Proben
Normales Gewebe
- 1–2 ml EDTA-Blut (empfohlene Probenart), oder
- Genomische DNA (1–2 µg)
Tumorgewebe
Tumorgehalt mindestens 20 %
- FFPE-Tumorblock (min. Gewebegröße 5x5x5 mm) (empfohlene Probenart)
- FFPE-Tumorgewebe-Objektträger (min. 10 Schnitte 4-10 µm, Gewebegröße 5×5 mm)
- Genomische DNA (> 200 ng)
- frisch gefrorenes Tumorgewebe
- 3x 10 ml cfDNA-Röhrchen für Liquid Biopsy
Hier finden Sie weitere Informationen zum sicheren Versand Ihrer Probe.
Weitere Probenmaterialien
Andere Probenmaterialien sind auf Anfrage möglich. Bitte beachten Sie: Bei unzureichender Probenqualität oder Tumorgehalt kann die Analyse fehlschlagen.
Wenn Sie mehr als eine Option für Tumorproben haben, wenden Sie sich bitte an unseren Diagnostik Support.
Wir unterstützen Sie gerne bei der Auswahl der optimalen Probe für Ihre Patientinnen und Patienten. Für höchste Genauigkeit benötigen wir Tumor- und Normalgewebe für unser somatisches Tumordiagnostik-Panel.
Das macht unseren CancerPrecision® Service besonders
Die Kenntnis über das molekulargenetische Tumorprofil ist für die Personalisierung der Behandlung und die Identifizierung zusätzlicher Behandlungsoptionen von entscheidender Bedeutung. Da die Anzahl der Biomarker, die wir in unserem CancerPrecision®-Befund adressieren, sehr hoch ist, werden die Ergebnisse in Hauptkategorien gruppiert und als Kacheln dargestellt. Behandlungsrelevante Ergebnisse sind farblich hervorgehoben.
Der Befund enthält einzelne Kacheln für Tumorgehalt, TMB, MSI, sowie den HRD-Score der analysierten Probe. Zudem enthält der Befund separate Kacheln für strukturelle Variantenbefunde, Tumortreibermutationen, Hinweise auf Virusinfektionen (HPV/EBV), Keimbahnvarianten (die die Erkrankung der Patientin oder des Patienten erklären bzw. therapierelevant sind), pharmakogenetische Varianten mit Auswirkungen auf die Tumorbehandlung und mögliche klonale Hämatopoese mit unbestimmtem Potenzial (CHIP).
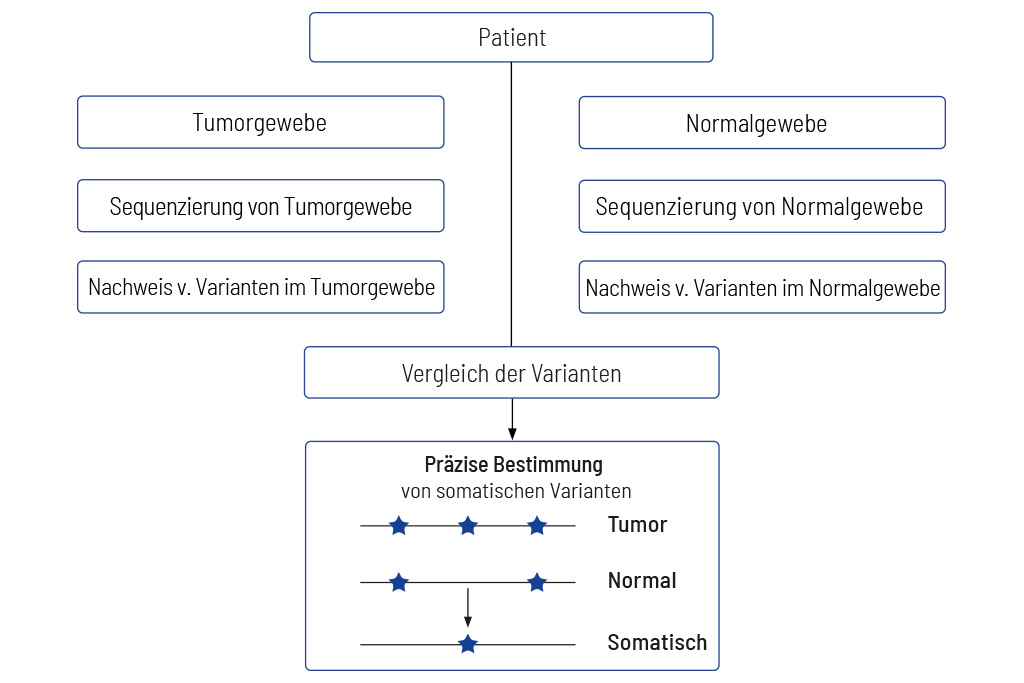
Vergleich von Tumor und Normalgewebe
Das einzig valide Vorgehen zur korrekten Ermittlung somatischer Varianten
Um aussagekräftige Ergebnisse zu erhalten, ist der Vergleich von Tumoren mit entsprechendem Normalgewebe unerlässlich. Diagnostische Tests, die keine Analyse von Tumoren und dazu passendem Normalgewebe durchführen, liefern in der Regel ungenaue Ergebnisse. Wir sequenzieren für unsere CancerPrecision®-Diagnostik immer Tumorgewebe und das entsprechende Normalgewebe.
In der Tumordiagnostik müssen Varianten unterschieden und verglichen werden, die auf den Tumor beschränkt sind (somatische Varianten) sowie Varianten, die auch im gesunden Gewebe vorhanden sind (Keimbahnvarianten).
Die einzig valide Methode, Varianten im gesunden Gewebe zu bestimmen, ist die Sequenzierung des passenden Normalgewebes zusammen mit dem Tumorgewebe. Methoden, die versuchen, die Normalgewebesequenzierung durch bioinformatische Ansätze zu ersetzen, können nicht klar zwischen Keimbahn- und somatischen Varianten unterscheiden, insbesondere wenn der Tumorgehalt der Probe hoch ist.1, 2 Ferner können diese Methoden, unter anderem, zu einer falschen Berechnung des TMBs führen, wodurch Patientinnen und Patienten im schlimmsten Fall nicht optimal behandelt werden.3
Aus diesem Grund sequenzieren wir immer sowohl DNA aus dem Tumor als auch aus dem Normalgewebe (meist Blut). Die Sequenzierungsdaten beider Gewebe werden verglichen und so die somatischen Varianten korrekt bestimmt.
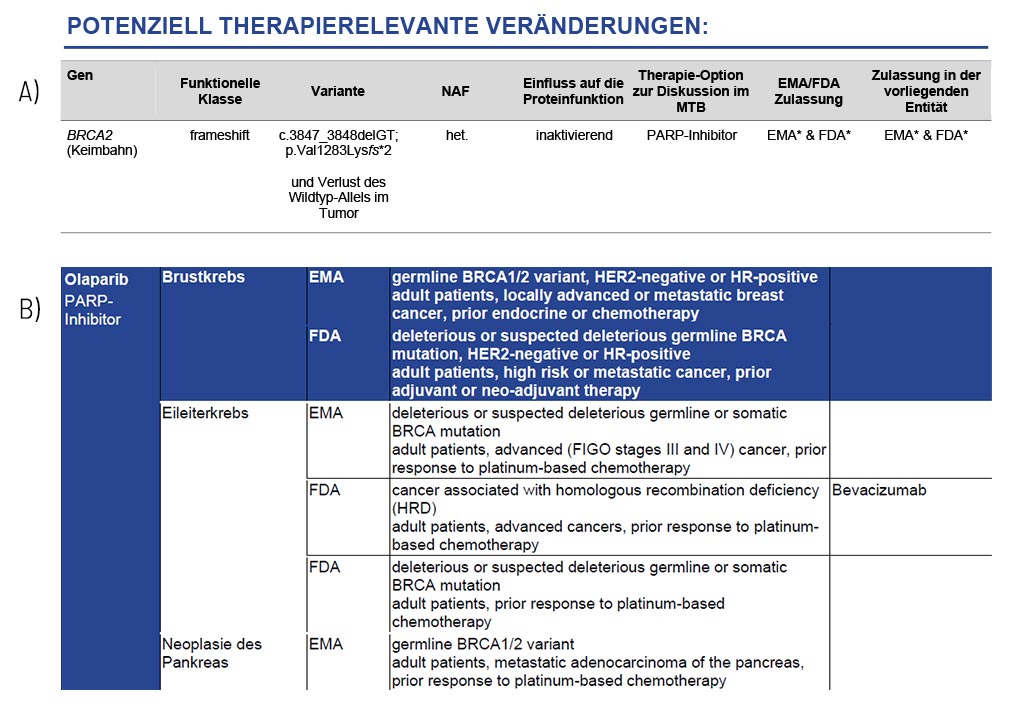
Varianten mit potenzieller therapeutischer Relevanz
Wegweiser für potenziell wirksame Medikamente
Die einzelnen therapierelevanten Veränderungen werden für jedes Gen detailliert dargestellt und die sich daraus ergebenden therapeutischen Optionen, einschließlich der EMA/FDA-Zulassung, angegeben (A). Diese Optionen bilden die Grundlage für die Diskussion in einem molekularen Tumorboard (MTB).
Im Anhang des medizinischen Befunds stellen wir eine umfangreiche Liste möglicher therapeutischer Strategien für jede identifizierte somatische Veränderung zur Verfügung (B). Diese Liste enthält neben den Medikamentenklassen und -namen auch deren Zulassung (FDA/EMA) und einschränkende Bedingungen.
Beispielbefund: Exemplarisch für die befundete BRCA2-Variante und die sich daraus ergebenden therapeutischen Optionen bei einer Brustkrebspatientin. Oberer Teil (A): Ein Ausschnitt aus Tabelle 1 des Befunds, in dem Varianten mit therapeutischer Relevanz aufgeführt sind. Unterer Teil (B): Ein Auszug der Medikamenten-Auflistung. Neben den abgebildeten Medikamenten werden auch andere Medikamente beschrieben.
Darstellung der Signalwege
Für ein detailliertes Verständnis der veränderten Signalkaskaden
Tumoren entstehen als Folge eines abweichenden Zellverhaltens bezüglich Zellwachstum und Zellsterben. Beide Prozesse geraten im Laufe der Tumorentwicklung außer Kontrolle. Typischerweise werden alle zellulären Prozesse durch ein komplexes Netzwerk von Signalwegen stark reguliert und kontrolliert.
Unser medizinischer Befund bietet einen umfassenden Überblick über das Netzwerk der tumorassoziierten Signalwege und ihre molekularen “Schlüsselakteure” sowie über alle relevanten genetischen Veränderungen und verfügbaren Medikamentenklassen, um:
- die Wechselwirkungen zwischen den verschiedenen Signalwegen zu verstehen und
- möglichen Umgehungsstrategien des Tumors entgegenzuwirken.
Tumoren zeichnen sich durch eine Anhäufung von Mutationen in Genen aus, die Schlüsselrollen in dieser komplexen Signalmaschinerie erfüllen. Eine einzige genetische Veränderung kann mehrere Signalwege beeinflussen. Daher ist es, neben dem Nachweis von krankheitsassoziierten Mutationen, entscheidend, das gesamte Zusammenspiel der Signalwege, die von den genetischen Varianten beeinflusst werden, zu verstehen. Dieser Ansatz hilft mögliche Ausweichstrategien des Tumors zu identifizieren. So können alle möglichen therapeutischen Optionen, einschließlich wirksamer Kombinationstherapien, in Betracht gezogen werden.
Berücksichtigte Signalwege
- Signalübertragung über Rezeptor-Tyrosinkinasen
- Zellzyklus
- Reparatur von DNA-Schäden
- Hormonelle Wege
- Wnt-Signalweg
- Hedgehog-Signalweg
- Hippo-Signalweg
- Apoptosis-Signalweg
- Epigenetische Regulatoren
TMB Bestimmung und MSI Vorhersage
Die Grundlage für therapeutische Entscheidungen über Immuntherapien mit Checkpoint-Inhibitoren
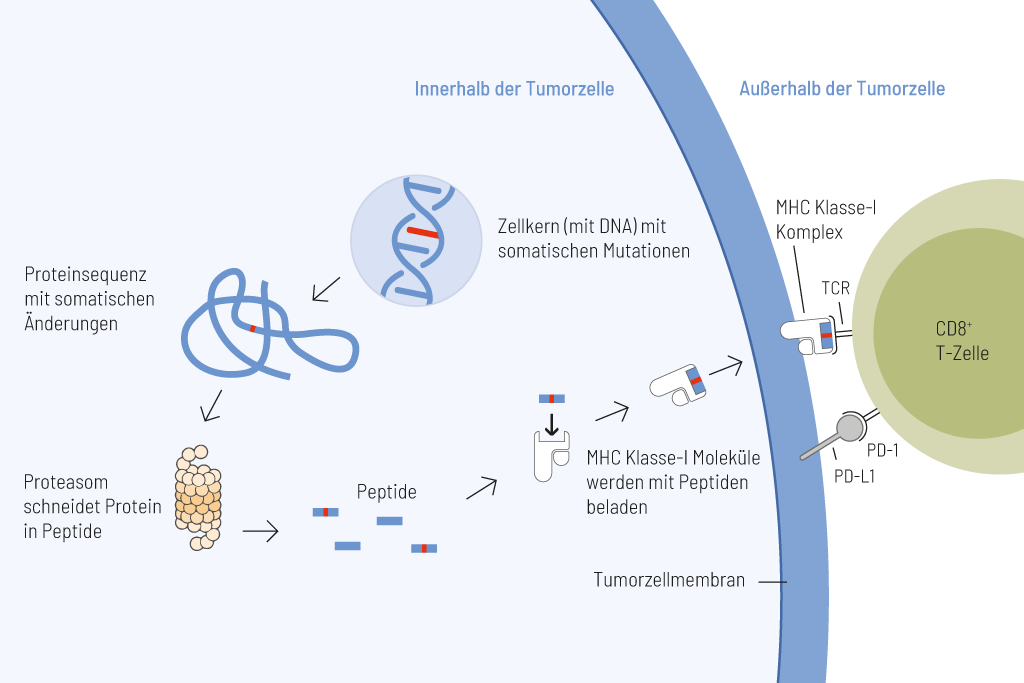
Präsentation von somatischen Peptiden, die von Tumorzellen stammen. Somatische Mutationen treten bei Krebs häufig auf und verändern dauerhaft die genomische Information. Diese genetischen Veränderungen können zur Expression von Proteinen mit veränderten Aminosäuresequenzen führen. Peptide, die eine somatische Veränderung tragen und damit ein besonders starkes immunstimulierendes Potenzial aufweisen, können auf der Oberfläche von Tumorzellen präsentiert werden und eine wirksame Anti-Tumor-Immunantwort hervorrufen.
Die Tumormutationslast (TMB), also die Anzahl der somatischen Mutationen pro Megabase (Mut/Mb), ist ein zuverlässiger Biomarker für das Ansprechen auf eine Behandlung mit Immun-Checkpoint-Inhibitoren.
Je höher die Anzahl der genetischen Veränderungen innerhalb einer Tumorzelle ist, desto mehr mutierte Proteine werden exprimiert. Diese mutierten Proteine werden zu kurzen Fragmenten (Peptiden) verarbeitet, die auf der Zelloberfläche von Tumorzellen präsentiert werden. Solche mutierten Peptide werden als Neoantigene bezeichnet und sind hoch immunogen. Das bedeutet, dass sie von Immunzellen, insbesondere von T-Zellen, sehr effektiv erkannt werden. T-Zellen sind in der Lage, Tumorzellen nach der Antigenerkennung direkt zu eliminieren. Je höher die Zahl der Mutationen ist, desto größer ist daher die Chance, dass Neoantigene auf Tumorzellen präsentiert werden, und desto effizienter ist die Tumorbekämpfung durch T-Zellen.
Mit der Analyse unserer umfassenden, hochsensitiven Panels im Tumor- und Normalgewebe, können wir den TMB präzise berechnen. Diese Metrik wird verwendet, um Tumoren in Gruppen mit niedriger und hoher Mutationslast zu klassifizieren. Wir führen die Klassifikation des TMBs sowie die genaue Mutationsrate der Tumorprobe auf.
Bei der Berechnung des TMBs ist auch die Größe des Panels für die Genauigkeit der Ergebnisse entscheidend. Mit einer Größe von 2,2 Mb liegt CancerPrecision® deutlich über der Mindestanforderung von 1,5 Mb und gewährleistet eine robuste Berechnung des TMBs.4
Die MSI (Mikrosatelliteninstabilität) ist ein weiterer wichtiger Parameter für das Ansprechen auf Immun- Checkpoint-Inhibitoren. Mikrosatelliten sind kleine repetitive DNA-Sequenzen, die sich im gesamten Genom finden. Die Größe von Mikrosatelliten kann sich aufgrund von Ausfällen der DNA-Mismatch Reparaturmechanismen verändern.
Traditionell wird der MSI-Status durch einen Vergleich von Mikrosatelliten-Regionen im Tumor- und Normalgewebe mittels PCR ermittelt. Wir sagen den MSI-Status durch NGS vorher. Diese Berechnung wurde mit Hunderten von Probenpaaren aus Normal- und Tumorgewebe verschiedener Tumorarten, bei denen mehr als 2.500 Mikrosatelliten-Foci untersucht wurden, validiert.
HRD – Homolge Rekombinationsdefizienz
Verschiedene DNA-Reperaturmechanismen sorgen in gesunden Zellen für ein stabiles und fehlerfreies Genom. Die homologe Rekombination (HR) ist hierbei für die Reparatur von DNA-Schäden, die beide DNA-Stränge betreffen (Doppelstrangbrüche), von besonderer Bedeutung. Ist dieser Mechanismus gestört sammeln sich Mutationen, Chromosomenaberrationen und andere Fehler im Genom an und man spricht von einer homologen Rekombinationsdefizienz (HRD). Durch die daraus resultierende genomische Instabilität begünstigt die HRD die Tumorentwicklung und trägt zur Tumorentstehung bei verschiedenen Tumorarten, insbesondere bei Brust- und Eierstockkrebs, bei.5, 6
Für zahlreiche HR-defiziente Tumoren gibt es wirksame therapeutische Ansätze, die sich die sogenannte synthetische Letalität zunutze machen, beispielsweise durch Verabreichung von PARP-Inhibitoren und platinhaltigen Chemotherapien. Diese Therapeutika verursachen DNA-Brüche, welche die verbleibende DNA-Reparaturmaschinerie von HR-defizienten Tumoren so stark belasten, dass diese absterben, während gesunde Zellen dank intakter Reperaturmechanismen überleben. Zur Identifizierung der Tumoren, bei denen diese Medikamente eingesetzt werden können, ist eine zuverlässige Bestimmung des HRD-Status von höchster Relevanz.
HR-defiziente Tumoren werden häufig durch Keimbahn oder somatische Mutationen in BRCA1 oder BRCA2 verursacht. Daher wurde dieses Muster früher als BRCAness bezeichnet. Darüber hinaus haben sich Mutationen in anderen HR-Genen wie RAD51C, ATM und PALB2 als mögliche Ursache für eine HRD erwiesen. Allerdings muss nicht jeder genetische Defekt in HR-Genen zwangsläufig zu einer HRD im Tumor führen. Zudem kann eine HRD auch dann vorliegen, wenn keine HR-Genmutation durch Sequenzierung nachweisbar ist (z. B. wurde eine Promotor-Methylierung von BRCAness-Genen ebenfalls als Ursache für eine HRD identifiziert). Werden ausschließlich Sequenz-Mutationen in BRCAness-Genen untersucht, kann eine potenzielle HRD unentdeckt bleiben. Um sicherzustellen, dass HR-defiziente Tumoren nicht übersehen werden, berechnen wir den HRD-Score als Teil jeder CancerPrecision®- Analyse – unabhängig von der Tumorentität.
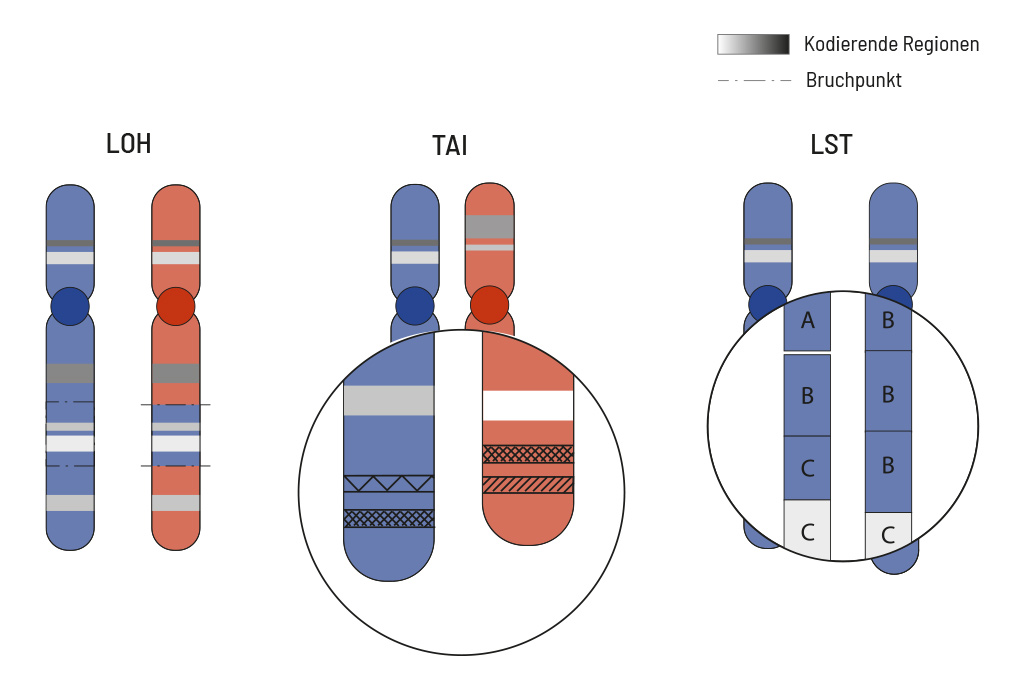
Der HRD-Score misst eine spezielle Signatur der genomischen Instabilität anhand der Anzahl der Chromosomenveränderungen wie Insertionen, Deletionen und Substitutionen, die auf genomweiter Ebene auftreten, auch ohne, dass die dafür verantwortlichen Genmutationen identifiziert werden müssen. Diese Signatur wird anschließend zur Berechnung des HRD-Scores der Tumorprobe verwendet. Der HRD-Score wird aus drei typischen HRD-Ereignissen berechnet:
- Loss of heterozygosity (LOH)
- Telomeric allelic imbalance (TAI)
- Large-scale state transition (LST)
Der HRD-Score wird in unserem CancerPrecision®- Befund zusammen mit allen identifizierten somatischen Mutationen und ausgewählten Genfusionen sowie TMB, MSI und CNVs angegeben.
LOH ist der irreversible Verlust eines elterlichen Allels, welcher besonders schwerwiegend ist, wenn das andere Allel bereits inaktiviert ist. LOH-Regionen werden definiert als Regionen, die größer als 15 Mb, aber kleiner als das gesamte Chromosom sind. Eine TAI tritt auf, wenn das Telomer eines Chromosoms in einem der beiden Chromosomen stark verkürzt ist, wodurch ein allelisches Ungleichgewicht in dieser Region entsteht. Dieses Ungleichgewicht resultiert daraus, dass die repetitiven DNA-Sequenzen in Telomerregionen besonders anfällig für HRD sind. Zur Bestimmung der LST wird die Anzahl der Bruchpunkte zwischen benachbarten Chromosomenregionen bewertet, die Kopienzahlgewinne oder -verluste von mehr als 10 Mb umfassen.
CNV-Analyse
Bestimmung von Deletionen/Amplifikationen für die höchste therapeutische Ausbeute
CNVs (Copy Number Variants) spielen in der Tumorgenetik oft eine wichtige Rolle. Die Kenntnis der Veränderungen bei CNVs hilft bei der Wahl der optimalen Behandlung. Die CNV-Analyse ist daher bei uns ein integraler Bestandteil der somatischen Tumordiagnostik.
Zelluläre Prozesse sind streng reguliert. Diese Regulierung hängt von der korrekten Funktion der Gene ab. Bei Tumoren ist die Kopienzahl der Gene häufig verändert, wodurch die korrekte Funktion der betroffenen Gene beeinträchtigt wird. Eine Erhöhung der Kopienzahl eines Gens kann dessen Aktivität steigern, während eine (teilweise) Deletion zu einem Funktionsverlust führen kann. Daher können Chromosomenaberrationen, die zu Veränderungen der Kopienzahl führen, auch therapeutische Konsequenzen haben.
Bei Tumoren sind Kopienzahlveränderungen (CNVs) aufgrund der allgemeinen genomischen Instabilität häufig. Hier sind oft große Chromosomenteile entweder deletiert oder amplifiziert. Es ist wichtig, diese Deletionen/Amplifikationen zu verstehen und die Gene in der betroffenen Region mit therapeutischer Relevanz zu kennen. Daher ermitteln wir Deletionen und Amplifikationen anhand der NGS-Daten.
Zusammen mit den betroffenen Genen mit therapeutischer Relevanz werden die Deletionen und Amplifikationen zu Beginn des Befunds aufgelistet. Ein vollständiges CNV-Profil der analysierten Regionen ist im Anhang des Befunds zu finden.
CancerFusionRx®
RNA-basierte Identifizierung von Fusionstranskripten
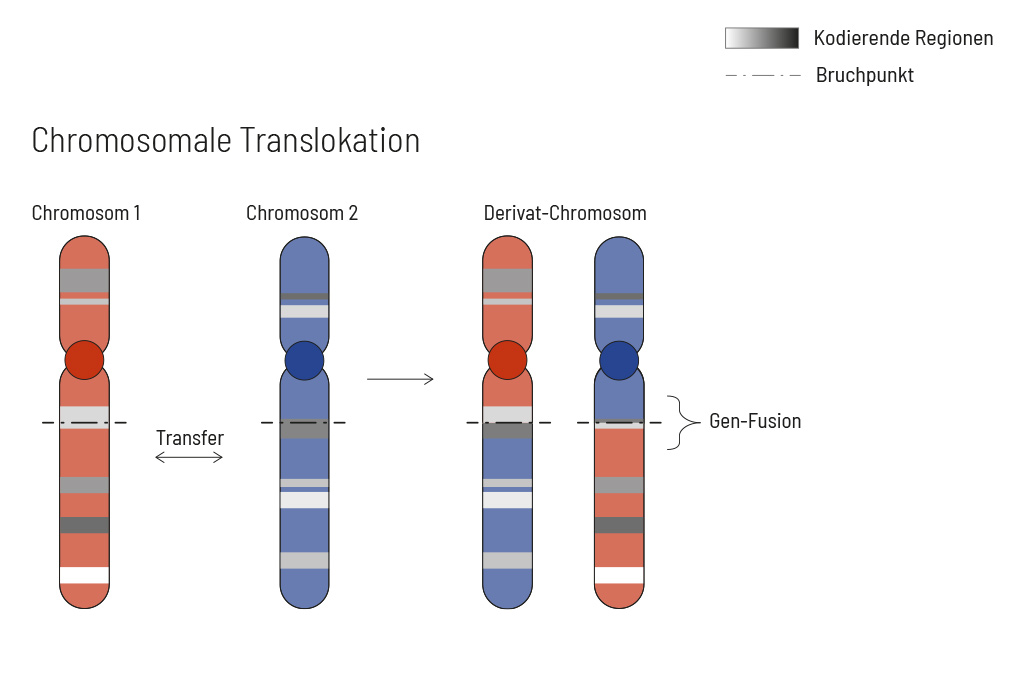
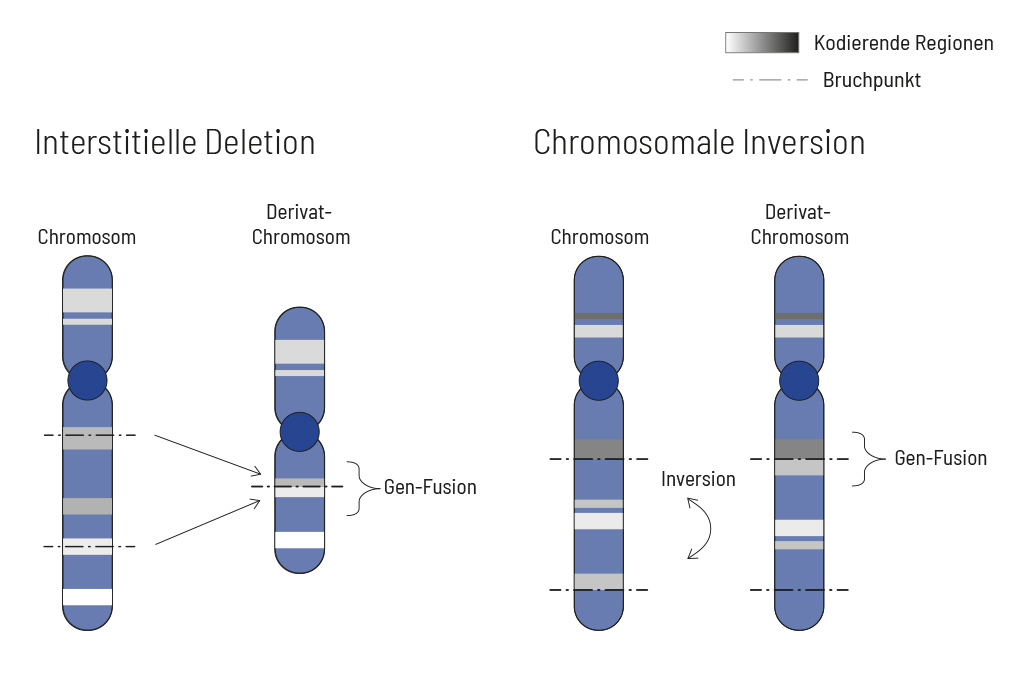
Chromosomale Veränderungen treten häufig und unabhängig von der Tumorentität auf. Infolgedessen kann es zu Genfusionen im Tumorgenom kommen. Fusionen sind oftmals starke Treiber der Tumorerkrankung und daher für Behandlungsentscheidungen sehr wichtig. Herkömmliche, auf PCR-Technologie basierende Methoden, können eine Fusion nicht nachweisen, wenn der Fusionspartner nicht bekannt ist (dies ist bei NTRK-Fusionen häufig der Fall). Selbst Analysen des gesamten Transkriptoms sind nicht sensitiv genug, insbesondere wenn der Tumorgehalt niedrig ist. Um alle bekannten und zuvor beschriebenen, sowie neuartigen Genfusionen mit einer therapeutischen Option nachzuweisen, haben wir eine gezielte Anreicherung auf RNA-Basis entwickelt.
Das Design umfasst aktuell mehr als 150 Gene für den Nachweis von Fusionen und beinhaltet über 120 Exon-Exon spezifische Anreicherungen mit bekannten Bruchpunkten. Diese Methode ist den DNA-basierten Methoden und auch Ansätzen auf Transkriptom-Basis überlegen. Wir empfehlen dringend, die genetische Tumordiagnostik durch RNA-Anreicherung für Fusionen zu ergänzen, um ein möglichst vollständiges Verständnis der Tumorbiologie zu erhalten.
Genverzeichnis
Genliste für DNA-basierte Analyse (749 Gene, CancerPrecision®, TUM01)
AAK1, ABCB1, ABCG2, ABL1, ABL2, ABRAXAS1, ACD, ACVR1, ADGRA2, ADRB1, ADRB2, AIP, AIRE, AJUBA, AKT1, AKT2, AKT3, ALK, ALOX12B, AMER1, ANKRD26, APC, APLNR, APOBEC3A, APOBEC3B, AR, ARAF, ARHGAP35, ARID1A, ARID1B, ARID2, ARID5B, ASXL1, ASXL2, ATM, ATR, ATRX, AURKA, AURKB, AURKC, AXIN1, AXIN2, AXL, B2M, BAP1, BARD1, BAX, BCHE, BCL10, BCL11A, BCL11B, BCL2, BCL3, BCL6, BCL9, BCL9L, BCOR, BCORL1, BCR, BIRC2, BIRC3, BIRC5, BLM, BMI1, BMPR1A, BRAF, BRCA1, BRCA2, BRD3, BRD4, BRD7, BRIP1, BTK, BUB1B, CALR, CAMK2G, CARD11, CASP8, CBFB, CBL, CBLB, CBLC, CCDC6, CCND1, CCND2, CCND3, CCNE1, CD274, CD79A, CD79B, CD82, CDC73, CDH1, CDH11, CDH2, CDH5, CDK1, CDK12, CDK4, CDK5, CDK6, CDK8, CDKN1A, CDKN1B, CDKN1C, CDKN2A, CDKN2B, CDKN2C, CEBPA, CENPA, CEP57, CFTR, CHD1, CHD2, CHD4, CHEK1, CHEK2, CIC, CIITA, CKS1B, CNKSR1, COL1A1, COMT, COQ2, CREB1, CREBBP, CRKL, CRLF2, CRTC1, CSF1R, CSF3R, CSMD1, CSNK1A1, CTCF, CTLA4, CTNNA1, CTNNB1, CTR9, CTRC, CUX1, CXCR4, CYLD, CYP1A2, CYP2A7, CYP2B6, CYP2C19, CYP2C8, CYP2C9, CYP2D6, CYP3A4, CYP3A5, CYP4F2, DAXX, DCC, DDB2, DDR1, DDR2, DDX11, DDX3X, DDX41, DEK, DHFR, DICER1, DIS3L2, DNMT1, DNMT3A, DOT1L, DPYD, E2F3, EBP, EED, EFL1, EGFR, EGLN1, EGLN2, EIF1AX, ELAC2, ELF3, EME1, EML4, EMSY, EP300, EPAS1, EPCAM, EPHA2, EPHA3, EPHB4, EPHB6, ERBB2, ERBB3, ERBB4, ERCC1, ERCC2, ERCC3, ERCC4, ERCC5, ERG, ERRFI1, ESR1, ESR2, ETNK1, ETV1, ETV4, ETV5, ETV6, EWSR1, EXO1, EXT1, EXT2, EZH1, EZH2, FAN1, FANCA, FANCB, FANCC, FANCD2, FANCE, FANCF, FANCG, FANCI, FANCL, FANCM, FAS, FAT1, FBXO11, FBXW7, FEN1, FES, FGF10, FGF14, FGF19, FGF2, FGF23, FGF3, FGF4, FGF5, FGF6, FGF9, FGFBP1, FGFR1, FGFR2, FGFR3, FGFR4, FH, FLCN, FLI1, FLT1, FLT3, FLT4, FOXA1, FOXE1, FOXL2, FOXO1, FOXP1, FOXQ1, FRK, FRS2, FUBP1, FUS, FYN, G6PD, GALNT12, GATA1, GATA2, GATA3, GATA4, GATA6, GGT1, GLI1, GLI2, GLI3, GNA11, GNA13, GNAQ, GNAS, GNB3, GPC3, GPER1, GREM1, GRIN2A, GRM3, GSK3A, GSK3B, GSTP1, H3-3A, H3-3B, H3C2, HABP2, HCK, HDAC1, HDAC2, HDAC6, HGF, HIF1A, HLA-A, HLA-B, HLA-C, HLA-DPA1, HLA, DPB1, HLA-DQA1, HLA-DQB1, HLA-DRA, HLA-DRB1, HMGA2, HMGCR, HMGN1, HNF1A, HNF1B, HOXB13, HRAS, HSD3B1, HSP90AA1, HSP90AB1, HTR2A, ID2, ID3, IDH1, IDH2, IDO1, IFNGR1, IFNGR2, IGF1R, IGF2, IGF2R, IKBKB, IKBKE, IKZF1, IKZF3, IL1B, IL1RN, ING4, INPP4A, INPP4B, INPPL1, INSR, IRF1, IRF2, IRS1, IRS2, ITPA, JAK1, JAK2, JAK3, JUN, KAT6A, KDM5A, KDM5C, KDM6A, KDR, KEAP1, KIAA1549, KIF1B, KIT, KLF2, KLF4, KLHL6, KLLN, KMT2A, KMT2B, KMT2C, KMT2D, KRAS, KSR1, LATS1, LATS2, LCK, LIG4, LIMK2, LRP1B, LRRK2, LTK, LYN, LZTR1, MAD2L2, MAF, MAGI1, MAGI2, MAML1, MAP2K1, MAP2K2, MAP2K3, MAP2K4, MAP2K5, MAP2K6, MAP2K7, MAP3K1, MAP3K13, MAP3K14, MAP3K3, MAP3K4, MAP3K6, MAP3K8, MAPK1, MAPK11, MAPK12, MAPK14, MAPK3, MAX, MBD1, MBD4, MC1R, MCL1, MDC1, MDH2, MDM2, MDM4, MECOM, MED12, MEF2B, MEN1, MERTK, MET, MGA, MGMT, MITF, MLH1, MLH3, MLLT10, MLLT3, MN1, MPL, MRE11, MS4A1, MSH2, MSH3, MSH4, MSH5, MSH6, MSR1, MST1R, MTAP, MTHFR, MTOR, MT-RNR1, MTRR, MUC1, MUTYH, MXI1, MYB, MYC, MYCL, MYCN, MYD88, MYH11, MYH9, NAT2, NBN, NCOA1, NCOA3, NCOR1, NF1, NF2, NFE2L2, NFKB1, NFKB2, NFKBIA, NFKBIE, NIN, NKX2-1, NLRC5, NOTCH1, NOTCH2, NOTCH3, NOTCH4, NPM1, NQO1, NR1I3, NRAS, NRG1, NSD1, NSD2, NSD3, NT5C2, NTHL1, NTRK1, NTRK2, NTRK3, NUMA1, NUP98, NUTM1, OBSCN, OPRM1, PAK1, PAK3, PAK4, PALB2, PALLD, PARP1, PARP2, PARP4, PAX3, PAX5, PAX7, PBK, PBRM1, PBX1, PDCD1, PDCD1LG2, PDGFA, PDGFB, PDGFC, PDGFD, PDGFRA, PDGFRB, PDK1, PDPK1, PGR, PHF6, PHOX2B, PIAS4, PIGA, PIK3C2A, PIK3C2B, PIK3C2G, PIK3CA, PIK3CB, PIK3CD, PIK3CG, PIK3R1, PIK3R2, PIK3R3, PIM1, PLCG1, PLCG2, PLK1, PML, PMS1, PMS2, POLD1, POLE, POLH, POLQ, POT1, PPM1D, PPP2R1A, PPP2R2A, PREX2, PRKAR1A, PRKCA, PRKCI, PRKDC, PRKN, PRMT5, PRSS1, PSMB1, PSMB10, PSMB2, PSMB5, PSMB8, PSMB9, PSMC3IP, PSME1, PSME2, PSME3, PSPH, PTCH1, PTCH2, PTEN, PTGS2, PTK2, PTK7, PTPN11, PTPN12, PTPRC, PTPRD, PTPRS, PTPRT, RABL3, RAC1, RAC2, RAD21, RAD50, RAD51, RAD51B, RAD51C, RAD51D, RAD54B, RAD54L, RAF1, RALGDS, RARA, RASA1, RASAL1, RB1, RBM10, RECQL4, REST, RET, RFC2, RFWD3, RFX5, RFXANK, RFXAP, RHBDF2, RHEB, RHOA, RICTOR, RINT1, RIPK1, RIT1, RNASEH2B, RNASEL, RNF43, ROS1, RPS20, RPS6KB1, RPS6KB2, RPTOR, RSF1, RUNX1, RYR1, SAMHD1, SAV1, SBDS, SCG5, SDHA, SDHAF2, SDHB, SDHC, SDHD, SEC23B, SERPINB9, SETBP1, SETD2, SETDB1, SF3B1, SGK1, SH2B1, SH2B3, SHH, SIK2, SIN3A, SKP2, SLC19A1, SLC26A3, SLCO1B1, SLIT2, SLX4, SMAD3, SMAD4, SMARCA4, SMARCB1, SMARCE1, SMC1A, SMC3, SMO, SOCS1, SOS1, SOX11, SOX2, SOX9, SPEN, SPINK1, SPOP, SPRED1, SRC, SRD5A2, SRGAP1, SRSF2, SSTR2, SSX1, STAG1, STAG2, STAT1, STAT3, STAT5A, STAT5B, STK11, SUFU, SUZ12, SYK, TAF1, TAF15, TAP1, TAP2, TAPBP, TBK1, TBL1XR1, TBX3, TCF3, TCF4, TCL1A, TEK, TERC, TERF2IP, TERT, TET1, TET2, TFE3, TGFB1, TGFBR2, TMEM127, TMPRSS2, TNFAIP3, TNFRSF13B, TNFRSF14, TNFRSF8, TNFSF11, TNK2, TOP1, TOP2A, TP53, TP53BP1, TP63, TPMT, TPX2, TRAF2, TRAF3, TRAF5, TRAF6, TRAF7, TRIM28, TRRAP, TSC1, TSC2, TSHR, TTK, TYMS, U2AF1, UBE2T, UBR5, UGT1A1, UGT2B15, UGT2B7, UIMC1, UNG, USP9X, VEGFA, VEGFB, VHL, VKORC1, WRN, WT1, XIAP, XPA, XPC, XPO1, XRCC1, XRCC2, XRCC3, XRCC5, XRCC6, YAP1, YES1, ZFHX3, ZNF217, ZNF703, ZNRF3, ZRSR2
DNA-basierte Detektion von ausgewählten strukturellen Veränderungen in den Genen
ALK, BCL2, BCR, BRAF, BRD4, EGFR, ERG, ETV4, ETV6, EWSR1, FGFR1, FGFR2, FGFR3, FUS, MET, MYB, MYC, NOTCH2, NTRK1, NTRK2, NTRK3, PAX3, PDGFB, RAF1, RARA, RET, ROS1, SSX1, SUZ12, TAF15, TCF3, TFE3, TMPRSS2
Genliste für RNA-basierte Fusionstranskriptanalyse (CancerFusionRx®, STR01)
Genliste für de-novo Fusionstranskripterkennung
ABL1, ACTB, AFAP1, AGK, AKAP12, AKAP4, AKAP9, AKT2, AKT3, ALK, ASPSCR1, ATF1, ATP1B1, ATRX, BAG4, BCL2, BCOR, BCORL1, BCR, BICC1, BRAF, BRD3, BRD4, c11orf95, CAMTA1, CCAR2, CCDC6, CCDC88A, CCNB3, CCND1, CD74, CIC, CLTC, CNTRL, COL1A1, CREB1, CREB3L1, CREB3L2, CRTC1, DDIT3, DNAJB1, EGFR, EML4, EPC1, ERBB2, ERBB4, ERG, ESR1, ETV1, ETV4, ETV5, ETV6, EWSR1, EZR, FEV, FGFR1, FGFR2, FGFR3, FLI1, FN1, FOXO1, FOXO4, FUS, GLI1, GOPC, GPR128, HMGA2, JAZF1, KIAA1549, KIF5B, LMNA, LPP, MAGI3, MAML1, MAML2, MAML3, MET, MGA, MGMT, MITF, MKL2, MYB, MYC, NAB2, NCOA1, NCOA2, NCOA4, NFIB, NOTCH2, NPM1, NR4A3, NRG1, NRG2, NSD3, NTRK1, NTRK2, NTRK3, NUTM1, PAX3, PAX7, PAX8, PDGFB, PDGFRA, PDGFRB, PHF1, PIK3CA, PLAG1, PML, POU5F1, PPARGC1A, PPP1CB, PRKACA, PRKAR1A, PTPRZ1, QKI, RAF1, RANBP2, RARA, RELA, RELCH, RET, ROS1, RREB1, RSPO2, RSPO3, SDC1, SDC4, SHTN1, SLC34A2, SND1, SQSTM1, SS18, SSX1, SSX2, SSX4, STAT6, STRN, SUZ12, TACC1, TACC3, TAF15, TCF12, TERT, TFE3, TFG, THADA, TMPRSS2, TPM3, TPR, TRIM24, TRIM33, TRIO, VGLL2, WT1, WWTR1, YAP1, YWHAE, ZMYM2, ZNF703
Genliste für ausgewählte Bruchpunkte in diesen Fusiongenen
AFAP1-NTRK2, ATP1B1-NRG1, BCOR-CCNB3, BRD3-NUTM1, BRD4-NUTM1, CCDC6–RET, CCDC88A-ALK, CD74-NRG1, CD74-ROS1, CLTC-ALK, DNAJB1-PRKACA, EGFR-PPARGC1A, EML4-ALK, ETV6-NTRK2, ETV6-NTRK3, EWSR1-ATF1, EWSR1-ERG, EWSR1-FLI1, EWSR1-WT1, EZR-ROS1, FGFR2-BICC1, FGFR1-TACC1, FGFR2-TACC3, FGFR3-TACC3, KIAA1549-BRAF, KIF5B-ALK, KIF5B–RET, MGA-NUTM1, NAB2-STAT6, NCOA4–RET, NPM1-ALK, NSD3-NUTM1, PAX3-FOXO1, PAX7-FOXO1, PPP1CB-ALK, PRKAR1A-RET, QKI-NTRK2, SDC4-NRG1, SDC4-ROS1, SLC34A2-ROS1, SND1-BRAF, SS18-SSX1, SS18-SSX2, TMPRSS2-ERG, TPM3-ALK, TPM3-NTRK1, TPM3-ROS1, TPR-NTRK1, TRIM24-BRAF, TRIM24-NTRK2, TRIM33-RET, TRIO-TERT
Liste für spezifische Transkriptvarianten
EGFR del ex2-3, EGFR del ex2-4, EGFR del ex2-14, EGFR del ex2-22 (mLEEK), EGFR del ex5-6, EGFR del ex6-7, EGFR del ex9, EGFR del ex9-10, EGFR del ex10, EGFR del ex12, EGFR del ex25-26, EGFR del ex25-27, EGFR del ex26-27, EGFR VII, EGFR VIII, MET ex14 skipping
Referenzen
1 Jones, S. et al. Personalized genomic analyses for cancer mutation discovery and interpretation. Science translational medicine 7, 283ra53; 10.1126/scitranslmed.aaa7161. (2015).
2 Sun, J. X. et al. A computational approach to distinguish somatic vs. germline origin of genomic alterations from deep sequencing of cancer specimens without a matched normal. PLoS computational biology 14, e1005965; 10.1371/journal.pcbi.1005965 (2018).
3 Nassar, A. H. et al. Ancestry-driven recalibration of tumor mutational burden and disparate clinical outcomes in response to immune checkpoint inhibitors. Cancer cell 40, 1161-1172.e5; 10.1016/j.ccell.2022.08.022 (2022).
4 Buchhalter, I. et al. Size matters: Dissecting key parameters for panel-based tumor mutational burden analysis. International journal of cancer 144, 848–858; 10.1002/ijc.31878 (2019).
5 Heeke, A. L. et al. Prevalence of Homologous Recombination-Related Gene Mutations Across Multiple Cancer Types. JCO precision oncology 2018; 10.1200/PO.17.00286 (2018).
6 Nguyen, L., W M Martens, J., van Hoeck, A. & Cuppen, E. Pan-cancer landscape of homologous recombination deficiency. Nature communications 11, 5584; 10.1038/s41467-020-19406-4 (2020).
Weitere Informationen
Webinar: Entdecken Sie die Möglichkeiten der modernen Tumordiagnostik
Genetische Tumordiagnostik kann Leben retten
Hinweis: Die Inhalte sind nur in englischer Sprache verfügbar.
Downloads
Application Note A Smarter Way to Fish for Fusions (EN) herunterladen
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.