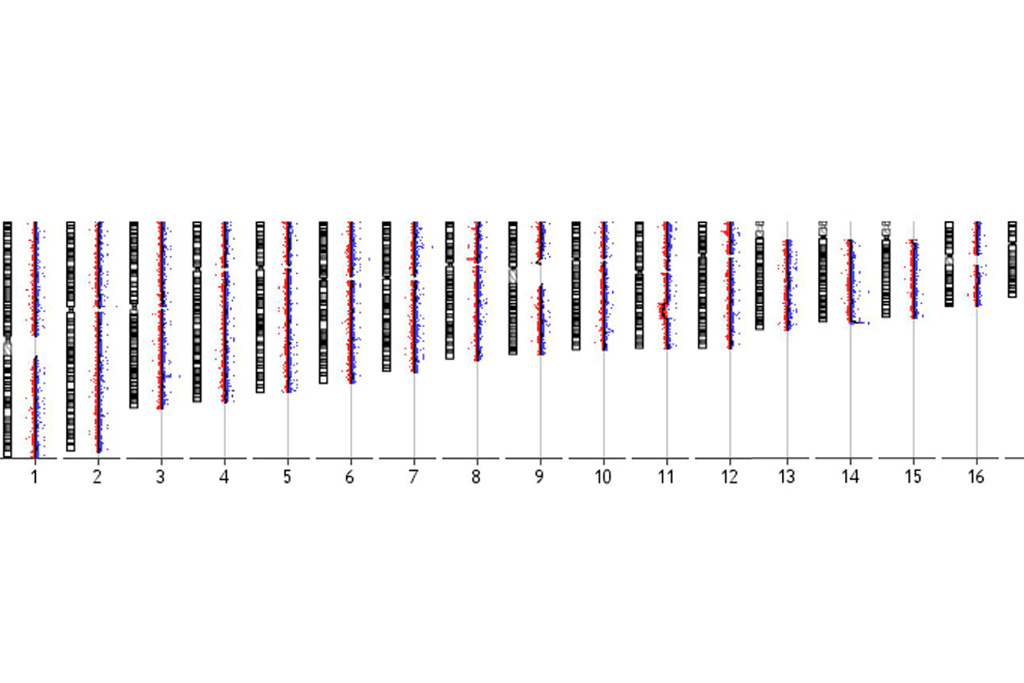
Die Array-CGH wird auch als Microarray-Analyse bezeichnet und stellt eine leistungsfähige methodische Weiterentwicklung der klassischen Zytogenetik dar. Mithilfe der Array-CGH können Verluste oder Zugewinne genomischer Bereiche in chromosomalem Material und damit der Diagnose unklarer Dysmorphie-, Retardierungs- und Fehlbildungssyndrome nachgewiesen werden. Im Vergleich zur konventionellen Chromosomenanalyse, bei der die Nachweisgrenze bei 5 bis 10 Mb liegt, erreicht die Microarray-Analyse einen Nachweis von genomischen Imbalancen mit einer diagnostischen Auflösung von 50 bis 100 kb.
Sie sind in Deutschland versichert? Unsere Kolleginnen und Kollegen vom Zentrum für Humangenetik Tübingen beraten Sie gerne!
Was wir Ihnen mit diesem Service bieten
Unser Versprechen an Sie
Anwendung von Array-CGH
Für viele diagnostische Fragestellungen stellt die Array-CGH inzwischen die „first-line“-Methode zur Abklärung der Ursache dar. Insbesondere ist der Einsatz der Array-CGH bei folgenden Fragestellungen indiziert:
- unklarer mentaler Retardierung (IQ < 70)
- Verdacht auf Veränderungen aus dem Autismus-Formenkreis
- Funktionsstörungen oder Fehlbildungen des Gehirns
- dysmorphologischen Auffälligkeiten
- angeborenen Fehlbildungen unklarer Äthiologie
- pränatale Ultraschallauffälligkeiten
- Wachstumsverzögerungen oder -auffälligkeiten (Prä- oder postnatal)
oder
- zur genaueren Charakterisierung bei zytogenetisch nachgewiesenen Aberrationen
- zur Überprüfung von balanziert erscheinenden Aberrationen bei klinisch auffälligen Patienten
- zur Charakterisierung von genomischen Rearrangements
- als ergänzende Untersuchung zu molekulargenetischen Verfahren (z. B. weiterführende Charakterisierung von MLPA-Befunden; gegebenenfalls Validierung von NGS-Ergebnissen)
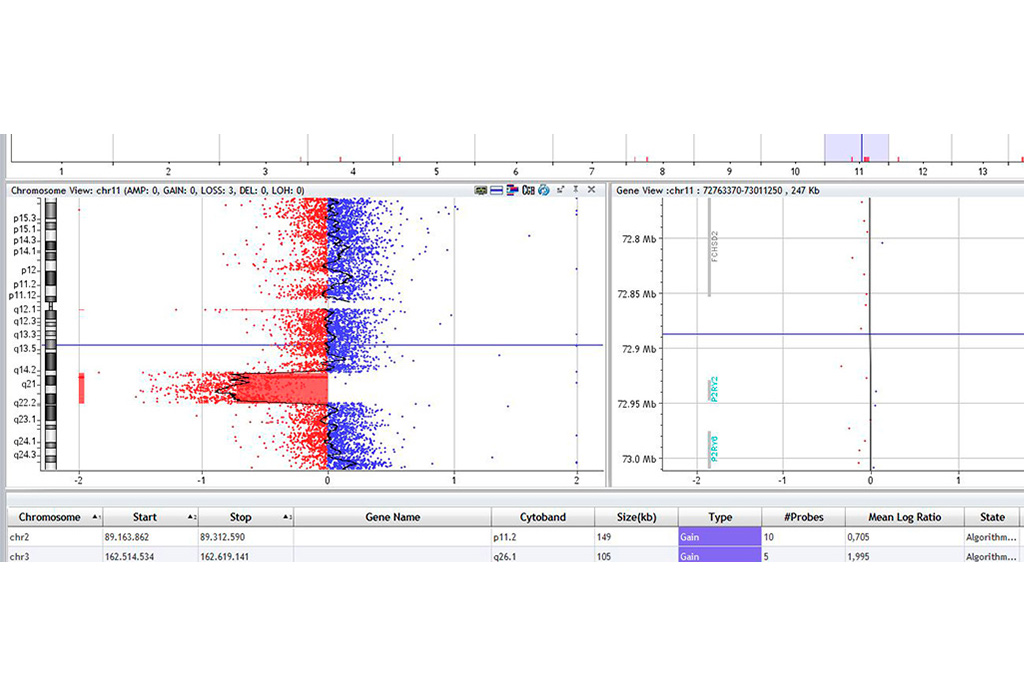
Beispielbefund
Allgemeine Informationen
Material
- 1 ml – 2 ml EDTA-Blut (empfohlene Probenart) oder 1 µg – 2 µg genomische DNA
- CeGaT Einsendeformular inkl. schriftliche Einverständniserklärung nach GenDG
Hier finden Sie weitere Informationen zum sicheren Versand Ihrer Probe.
Dauer
- Dauer der Untersuchung: 4-6 Wochen
Was ist eine Array-CGH?
Als Array-CGH-Analyse wird eine molekulargenetische Methode bezeichnet, mit deren Hilfe kleine Gewinne oder Verluste in chromosomalem Material nachgewiesen werden können, die unterhalb der Detektionsgrenze einer konventionellen Chromosomenanalyse liegen. Bei der Array-CGH (Array-based Comparative Genomic Hybridization) werden die Patienten-DNA und eine Referenz-DNA mit unterschiedlichen Fluoreszenz-Farbstoffen markiert und auf einem Trägerchip mit immobilisierten DNA-Fragmenten („Probes“ oder Sonden) cohybridisiert. Diese Sonden decken das menschliche Genom möglichst gleichmäßig ab. Numerische Veränderungen führen zu einer Farbverschiebung des Fluoreszenzsignals für die einzelnen Sonden. Diese Signale werden mittels eines Laserscanners erfasst und mit einer entsprechenden Software der jeweiligen chromosomalen Region zugeordnet und dargestellt („molekulares Karyotyping“).
Bei CeGaT können alle erhältlichen Arrayformate der Firma Agilent prozessiert, analysiert und befundet werden. In der Routine verwenden wir den Agilent 180K Array mit einer diagnostischen Auflösung von ≥ 50kb.
Nach Rücksprache besteht außerdem die Möglichkeit, individuelle hochauflösendere Arrays zu analysieren.
Präzise Ergebnisse durch die Array-CGH-Analyse
Durch die Detektion von Mikrodeletionen und Mikroduplikationen im Genom einer Patientin oder eines Patienten können mithilfe der Array-CGH-Analyse deutlich kleinere Abweichungen, als bei der konventionellen Chromosomenanalyse erkannt werden. Eine herkömmliche Chromosomenanalyse ermittelt nur bei etwa 3 %–4 % der untersuchten Patientinnen und Patienten mit Entwicklungsverzögerungen eine strukturelle genomische Veränderung. Durch die genomweite Analyse können mithilfe der Array-CGH-Analyse bei bis zu 15 % der Patientinnen und Patienten ursächliche Veränderungen detektiert werden. Die Array-CGH-Analyse kann unklare Fälle mentaler Retardierung oder seltene syndromale Krankheitsbilder auflösen. Außerdem können Verluste oder Zugewinne, die bei einer Chromosomenanalyse ermittelt wurden, präziser charakterisiert werden.
Downloads
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.